Künstliche Intelligenz als Schlüssel zur Erkennung von Gehirnarterien
In der medizinischen Bildgebung eröffnet Künstliche Intelligenz neue Wege: Durch fortschrittliche Algorithmen können nun auch in nicht-angiographischen Aufnahmen Gehirnarterien identifiziert und annotiert werden, was eine breitere Diagnose und Behandlung zerebraler Gefäßerkrankungen ermöglicht.
von Bertram Sabrowsky-Hirsch, MSc
Inhalt
- Innovation in der medizinischen Bildgebung: KI entdeckt Gehirnarterien
- Entschlüsselung des Circle of Willis: Vorbeugung neurologischer Krisen durch Bildgebung
- Durchblick im Gewebe-Dschungel: MR-Bildgebung enthüllt Blutgefäße
- Methodik der Zukunft: KI-gestützte Segmentierung der Gehirnarterien
- Ergebnisse der innovativen Gefäßsegmentierung in MR-Sequenzen durch KI-Methodik
- Fazit: Durchbruch in der Annotation zerebraler Arterien mit KI
- Danksagung
- Literaturverweise
- Autor

Innovation in der bildbasierten Diagnose: KI entdeckt Gehirnarterien
Stellen Sie sich vor, eine Technologie könnte das Risiko eines Schlaganfalls erkennen, bevor es zu spät ist. Weltweit fordert der Schlaganfall als zweithäufigste Todesursache zahllose Leben und hinterlässt bei Überlebenden oft schwere neurologische Schäden [1]. In Deutschland hatte einer von 40 Erwachsenen bereits einen Schlaganfall [2]. Hier spielt die neurologische Bildgebung eine kritische Rolle – sie ist der Schlüssel zur Diagnose und Behandlung dieser verhängnisvollen Erkrankungen.
Doch die Herausforderung: Blutgefäße sind auf herkömmlichen Bildern nur schwer zu erkennen. Traditionell setzt die Medizin auf angiographische Methoden, die zwar effektiv, aber nicht immer verfügbar sind und oft den Einsatz von Kontrastmitteln erfordern. Dies ist nicht nur kostspielig, sondern kann auch Risiken für die Patient*innen bergen.
Hier setzt die bahnbrechende Arbeit der RISC Software GmbH an. In der Publikation „Segmentation and Anatomical Annotation of Cerebral Arteries in Non-Angiographic MRI“ ergründen die Forscher*innen, wie künstliche Intelligenz dazu beitragen kann, Blutgefäße in Standard-MR-Bildern sichtbar zu machen – eine Revolution in der Bildgebung, die den Weg für frühere und breitere Diagnosen ebnet. Mit dieser innovativen Methode, die auch ohne spezielle angiographische Bilddaten funktioniert, eröffnet sich ein neues Feld für Begleitdiagnosen und ermöglicht es, historische Daten neu zu bewerten. In Zukunft könnte KI dazu beitragen Schlaganfälle zu verhindern.
Untersuchung des Circle of Willis: Vorbeugung neurologischer Notfälle durch Bildgebung
Direkt unterhalb des Gehirns befindet sich eine lebenswichtige arterielle Struktur, bekannt als der Circle of Willis (CoW). Seine uneingeschränkte Funktion ist für die Vermeidung neurologischer Notfälle unerlässlich, denn Störungen können zu gravierenden gesundheitlichen Konsequenzen führen. Moderne bildgebende Verfahren eröffnen neue Möglichkeiten, die verborgenen Aspekte dieser zentralen Gefäßstruktur zu beurteilen, was eine Früherkennung und Prävention von neurologischen Ereignissen ermöglicht.
Durch hochauflösende Scans können Mediziner*innen die komplexen Verzweigungen und Verbindungen des CoW detailliert betrachten und pathologische Zustände wie Aneurysmen, Stenosen, Verschlüsse und vaskuläre Malformationen aufspüren, die in diesem Gebiet häufiger vorkommen. Diese wertvollen Einblicke in die zerebrale Zirkulation sind der Schlüssel zur Vorbeugung von Schlaganfällen und anderen lebensbedrohlichen Komplikationen, indem sie rechtzeitige therapeutische Maßnahmen ermöglichen und die Gesundheit der Patient*innen zu wahren.
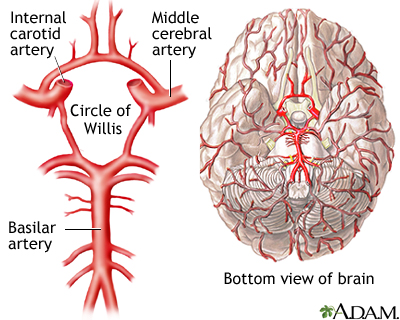
Abbildung 1: Der Arterienring des Circle of Willis versorgt das Gehirn mit Blut.
(Quelle: MedlinePlus, https://medlineplus.gov/ency/imagepages/18009.htm)
Durchblick im Gewebe-Dschungel: MR-Bildgebung enthüllt Blutgefäße
Wie einfach sich Blutgefäße in den Bilddaten von den umgebenden Strukturen abgrenzen lassen, hängt in erster Linie von der verwendeten Bildgebungsmethode ab. Die Magnetresonanzbildgebung (MR) bietet den Vorteil, dass sie nicht-invasiv ist und ohne den Einsatz von Röntgenstrahlen auskommt. Zudem stehen den Radiolog*innen unterschiedliche Sequenzen zur Verfügung, um verschiedene Gewebeeigenschaften zu betonen und so bestimmte anatomische Strukturen hervorzuheben. Die Time-of-Flight (TOF) Sequenz ermöglicht etwa den Blutfluss innerhalb der Arterien abzubilden, ist aber aufgrund des schwachen Kontrasts schlechter für die Beurteilung von Gehirngewebe geeignet als vergleichsweise die T2 Sequenz. Welche Sequenzen bei der Bildaufnahme zum Einsatz kommen, ist abhängig von der medizinischen Indikation und die Anzahl an verschiedenen Sequenzen wird zugunsten der Aufnahmedauer eingeschränkt. Bilddaten sind deshalb oft nicht verfügbar, was eine Beurteilung der Blutgefäße erschwert oder gar unmöglich macht. Abbildung 2 veranschaulicht die Sichtbarkeit von Blutgefäßen in unterschiedlichen MR-Sequenzen.
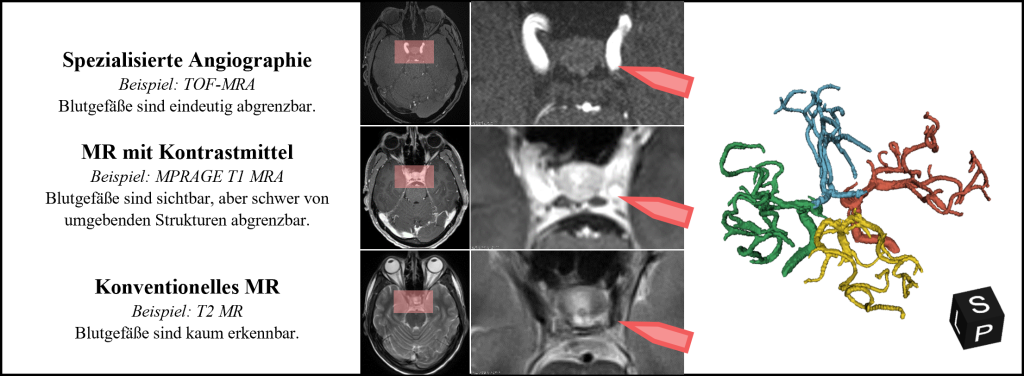
Abbildung 2: Die Sichtbarkeit von Blutgefäßen hängt stark von der verwendeten Bildgebungsmethode ab. In den Schnittbildern wird der gleiche Ausschnitt eines zerebralen Gefäßbaum (rechts) in drei unterschiedlichen Sequenzen abgebildet.
Methodik der Zukunft: KI-gestützte Segmentierung der Gehirnarterien
Die Forschenden der Abteilung Medizin-Informatik haben eine vollständig automatisierte Methode entwickelt, die die Gehirnarterien entlang des Circle of Willis segmentiert und vier Gefäßregionen (Anterior, Posterior, Links und Rechts) annotiert. Ihre Methode unterstützt drei verschiedene nicht-angiographische MR-Sequenzen sowie die angiographische TOF-MRA Sequenz. Hierfür wurde eine künstliche Intelligenz auf 555 Patient*innen des öffentlichen IXI-Datensatzes trainiert [3]. Für jeden Patient*in sind im besagten Datensatz die MR-Sequenzen PD, T1, T2 und TOF-MRA verfügbar. Die Segmentierungsaufgabe bei den angiographischen TOF-MRA Bilddaten konnte mit traditionellen Bildverarbeitungsmethoden automatisiert gelöst werden (siehe Abbildung 3), und die Annotationen wurden anschließend erfolgreich auf die nicht-angiographischen Sequenzen übertragen (siehe Abbildung 4). Die künstliche Intelligenz wurde daraufhin trainiert, die Segmentierungsaufgabe für alle vorhandenen Sequenzen zu meistern. Spezifisch wurden semantische Segmentierungsmodelle basierend auf künstlichen neuronalen Netzwerken (ANN) unter Verwendung des Frameworks nnU-Net entwickelt [4].

Abbildung 3: Die automatische Methode zur Annotation der Blutgefäße und Gefäßregionen in der angiographischen TOF-MRA Sequenz basiert auf traditionellen Bildverarbeitungsmethoden.
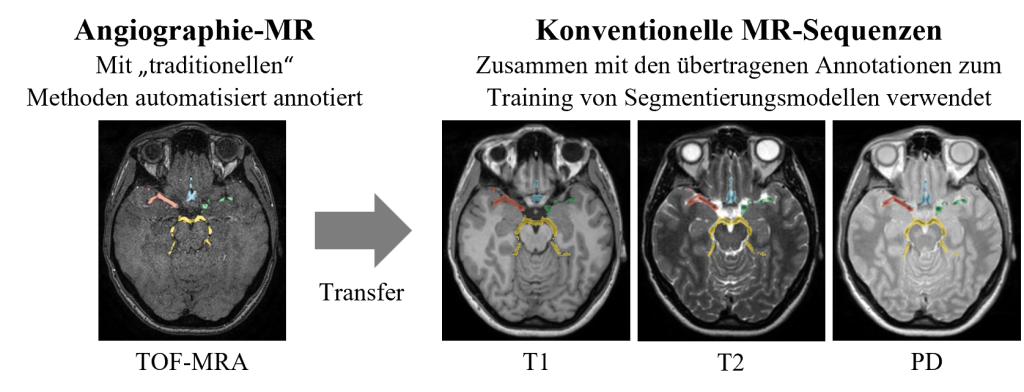
Abbildung 4: Die annotierten Blutgefäße aus der TOF-MRA Sequenz wurden auf die nicht-angiographischen Sequenzen (PD, T1, T2) übertragen. Der resultierende Datensatz aus Bilddaten und „Ground-Truth“ Annotationen wurde anschließend für das Training der Segmentierungsmodelle verwendet.
Ergebnisse der innovativen Gefäßsegmentierung in MR-Sequenzen durch KI-Methodik
Die Forschenden der Abteilung Medizin-Informatik führten das Training und die Auswertung ihrer Methode mittels einer 5-fachen Kreuzvalidierung über 485 Patientinnen durch. Zusätzlich wurden 70 der ursprünglichen 555 Patientinnen als Testdatensatz ausgewertet, die an einem anderen Institut mit abweichender Bildgebungshardware untersucht wurden. Diese Strategie diente der Überprüfung der Anwendbarkeit der Methode über verschiedene Institute hinweg.
Die Methode wurde von den Forschenden durch visuelle Begutachtung sowie quantitative Auswertung von Ähnlichkeitsmaßen auf der Basis der erzeugten Annotationen evaluiert. Hierfür wurden etablierte Metriken wie der Dice Similarity Coefficient (DSC), die 95 Perzentile der Hausdorff-Distanz (HD95) und die mittlere symmetrische Oberflächendistanz (ASD) berechnet. In der Kreuzvalidierung erreichte der DSC um den Circle of Willis einen sehr guten Wert von 0.82 (in der Literatur wird ein Wert von 0.7 als qualitativ hochwertig gewertet [5]). Die ASD-Metrik zeigte eine Abweichung von 0.44 mm, was in Anbetracht der durchschnittlichen Auflösung der Bilddaten von 0.92 mm als sehr gutes Ergebnis anzusehen ist. Die Ergebnisse deuten darauf hin, dass Gefäßstrukturen in den untersuchten MR-Sequenzen mit angemessener Qualität segmentiert und annotiert werden können.
Für den zusätzlichen Testdatensatz erzielten die Modelle einen DSC von 0.75, was trotz der deutlichen Reduktion immer noch ein gutes Ergebnis darstellt. Dies weist allerdings darauf hin, dass die Methode noch durch ein Training mit umfangreicheren Datensätzen aus verschiedenen Instituten verbessert werden sollte, um robustere Leistungen bei unterschiedlicher Bildgebungshardware zu erzielen.
Bei den einzelnen MR-Sequenzen waren deutliche Unterschiede zu verzeichnen: Die angiographische TOF-MRA Sequenz lieferte zwar die besten Ergebnisse, und die Segmentierungsaufgabe war hier durch die klare Sichtbarkeit der Blutgefäße aber auch relativ einfach. Basierend auf den Ergebnissen eignete sich die PD-Sequenz (gewichtet nach Protonendichte) am besten für die Annotation, gefolgt von T2 und T1. Auch bezüglich der Gefäßregionen wurden geringe Unterschiede beobachtet, wobei die Ergebnisse für die linke und rechte Region besser waren als für die anteriore und posteriore Region.
In der Literatur fehlen vergleichbare Studien, die auf konventionellen, nicht-angiographischen Bilddaten basieren. Klimont et al. [6] beschrieben eine Methode für nicht-angiographische CT-Bilddaten, jedoch nicht für MR-Sequenzen. Die Ergebnisse der Forschenden für die TOF-MRA Sequenz sind vergleichbar mit Werten aus der Literatur [7] und bieten durch die Unterstützung nicht-angiographischer MR-Sequenzen einen einzigartigen Vorteil.
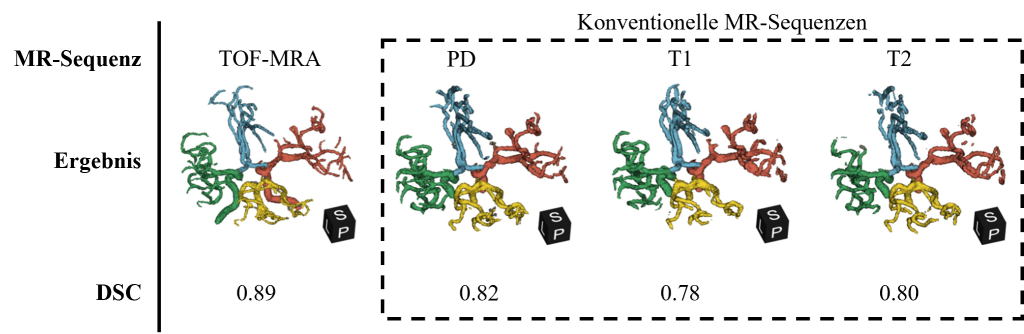
Abbildung 5: Visuelle Darstellung der Ergebnisse anhand eines Patienten aus den Kreuzvalidierungsdaten im Vergleich zu den verschiedenen MR-Sequenzen. Die DSC-Metrik bezieht sich auf den durchschnittlichen Wert im Bereich des Circle of Willis für die jeweilige Sequenz aus der Kreuzvalidierung.
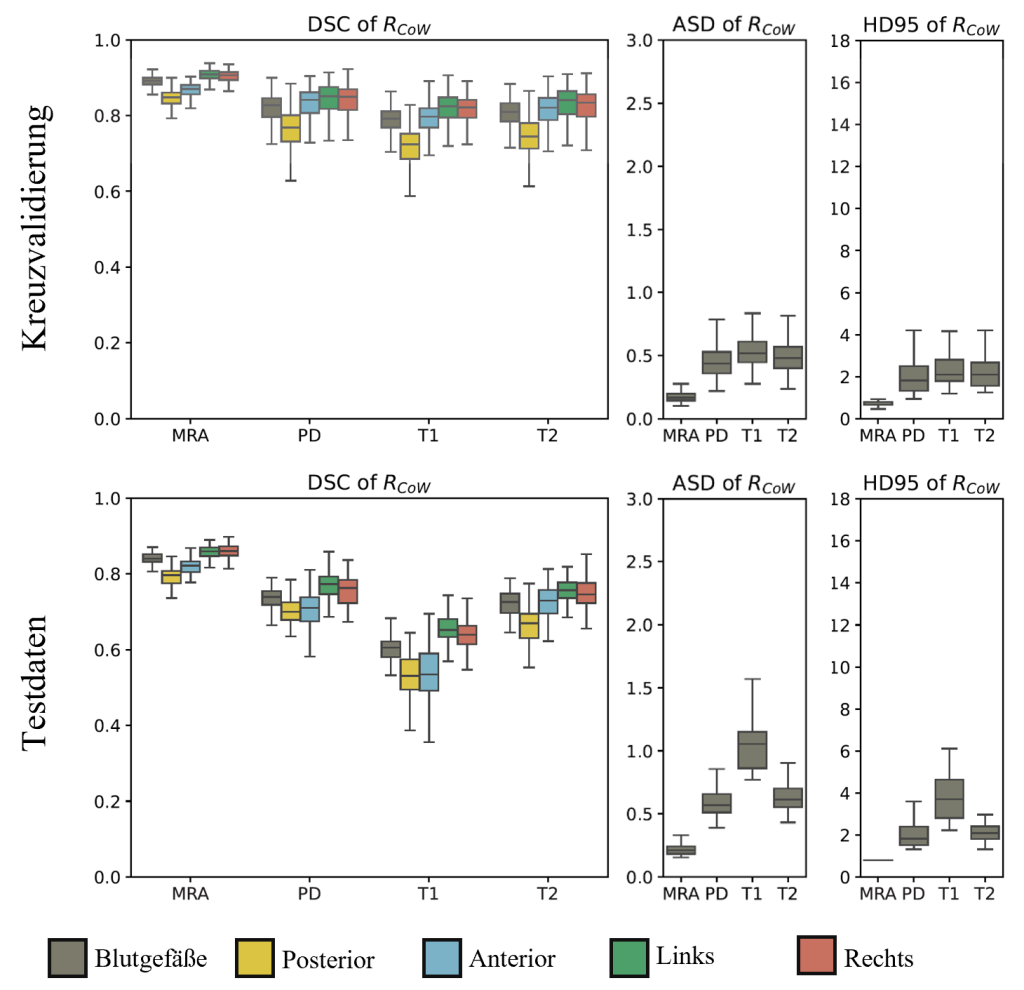
Abbildung 6: Detaillierte Auswertung der Metriken DSC, ASD und HD95 im Bereich des Circle of Willis im Rahmen der Kreuzvalidierung (oben) und für die zusätzlichen Testdaten (unten). Die Blutgefäße wurden in einem allgemeinen Label und zusätzlich für jede Gefäßregion (Posterior, Anterior, Links, Rechts) ausgewertet. Die Ergebnisse beziehen sich auf die jeweilige MR-Sequenz: (TOF-)MRA, PD, T1 und T2.
Fazit: Durchbruch in der Annotation zerebraler Arterien mit KI
Die Forschenden der Abteilung Medizin-Informatik haben in ihren Experimenten demonstriert, dass mit Hilfe künstlicher Intelligenz Blutgefäße auch in nicht-angiographischen Bilddaten qualitativ hochwertig annotiert werden können. Dies stellt einen bedeutenden Fortschritt im Bereich der Analyse medizinischer Bilddaten dar, da bisherige Methoden sich hauptsächlich auf angiographische Bilddaten fokussierten. Für eine breitere Anwendbarkeit der Methode ist eine Erweiterung der Trainingsdaten sowohl in Umfang als auch in der Variabilität bezüglich Bildgebungshardware und medizinischer Indikationen notwendig. Besonders die Einbeziehung von Daten von Patienten mit Aneurysmen und Tumoren ist entscheidend für eine robuste Segmentierung dieser Malformationen. Aktuell arbeiten die Forschenden weiterhin an der Unterstützung zusätzlicher MR-Sequenzen wie der MPRAGE MRA Sequenz.
Derzeit empfehlen die Forschenden den Einsatz ihrer Methode primär zur Lokalisierung des Circle of Willis und nicht zur Beurteilung der Gefäßstrukturen, da die korrekte Annotation von Malformationen noch nicht überprüft werden kann. Die Forschenden verwenden die erstellten Annotationen zurzeit der Lokalisierung und der automatischen Registrierung der Blutgefäße mit DSA (Digital Subtracted Angiography) Bilddaten. Geplante Erweiterungen könnten die Methode zukünftig jedoch auch für die Analyse von Blutgefäßen in nicht-angiographischen Bilddaten nutzbar machen.
Die Ergebnisse und die Methodik wurden auf der DMIP-Konferenz 2023 in Japan vorgestellt und sind in den Proceedings der ACM Digital Library zugänglich (ISBN: 979-8-4007-0942-5). Der annotierte Datensatz sowie die trainierten Modelle sind öffentlich im GitHub Repository der Abteilung einsehbar.
Link zum Repository: https://github.com/risc-mi/cerebral-artery-annotation.
Danksagung
Diese Arbeit wurde durch die FFG (Österreichische Forschungsförderungsgesellschaft) im Rahmen des Grants 872604 (MEDUSA) sowie durch Forschungsförderungsmitteln des Landes Oberösterreich finanziert. Die RISC Software GmbH ist Teil des Netzwerkes der UAR (Upper Austrian Research).
Literaturverweise
[1] Katan, M. and Luft, A. (2018). Global Burden of Stroke. Semin Neurol., 38(2), 208–211. https://doi.org/10.1055/s-0038-1649503
[2] Robert Koch-Institut (Hrsg) (2015) Gesundheit in Deutschland. Gesundheitsberichterstattung des Bundes. Gemeinsam getragen von RKI und Destatis. RKI, Berlin, https://doi.org/10.17886/rkipubl-2015-003-2
[3] Information eXtraction from Images (IXI) Dataset. (Retrieved March 23, 2023) from https://brain-development.org/ixi-dataset/
[4] Isensee, F., Jaeger, P. F., Kohl, S. A. A., Petersen, J., & Maier-Hein, K. H. (2021). nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation. Nat Methods, 18(2), 203–211. https://doi.org/10.1038/s41592-020-01008-z
[5] Zou, K. H., Warfield, S. K., Bharatha, A., et al. (2004). Statistical validation of image segmentation quality based on a spatial overlap index. Acad Radiol, 11(2), 178–189. https://doi.org/10.1016/s1076-6332(03)00671-8
[6] Klimont, M., et al. (2020). Deep learning for cerebral angiography segmentation from non-contrast computed tomography. PLOS ONE, 15(7), e0237092. https://doi.org/10.1371/journal.pone.0237092
[7] Hilbert, A., et al. (2020). BRAVE-NET: Fully Automated Arterial Brain Vessel Segmentation in Patients With Cerebrovascular Disease. Frontiers in Artificial Intelligence, 3. https://doi.org/10.3389/frai.2020.552258
Ansprechperson
Autor

Bertram Sabrowsky-Hirsch, MSc
Researcher & Developer