Medical Image Processing, Modeling and Simulation based on Artificial Intelligence
Der Forschungsbereich „Medical Image Processing, Modeling and Simulation based on Artificial Intelligence“ (MIMAS.ai) umfasst hochdynamische Forschungsthemen. Diese gewinnen nicht zuletzt durch aktuelle technologische Fortschritte in der Medizin stark an Bedeutung.
Medizinische Bildanalyse und Bildsegmentierung
Medizinische Bilddaten dienen der Diagnose, Behandlungsplanung, Begleitung von Eingriffen, Beobachtung von Zustandsveränderungen und Dokumentation. Die Bandbreite reicht von 2D-Bildern (z. B. Röntgen, Wundfotos) über 3D-Scans (CT, MRT, DSA) bis hin zu Videos (2D+t, 3D+t), die auch zeitliche Abläufe erfassen.
Um diese multimodalen Bilddaten nutzbar zu machen, entwickelt die Forschungsabteilung Medizin-Informatik Methoden zur Bildanalyse und Segmentierung. KI-basierte Verfahren registrieren (überlagern) die Daten und extrahieren patient*innenspezifische Strukturen wie Blutgefäße, Gewebe oder Haut. Diese segmentierten Strukturen bilden die Grundlage für medizinische Modelle und Simulationen.
Der Erfolg von Machine-Learning-Methoden hängt allerdings von der Qualität und Anzahl der Trainingsdaten ab. Gerade in der Medizin fehlen oft Daten mit „Ground Truth“, oder sie stehen aus Datenschutzgründen nicht zur Verfügung. Deshalb arbeitet das Team an Methoden zur vereinfachten, interaktiven Erstellung von Ground Truth. Dazu zählen z. B. CycleGANs zum Generieren synthetischer Trainingsdaten, One-Shot Learning zur Datenaugmentierung, Transfer Learning zur Wiederverwendung bestehender Wissensmodelle sowie Domain Adaptation zur Anpassung an neue Datenverteilungen.
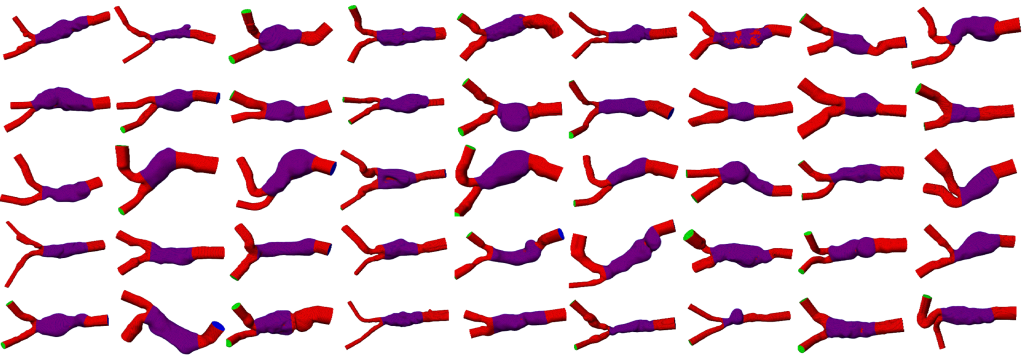
Deep Learning gestützte Extraktion anatomischer Strukturen für Patient*innen-Kohorten am Beispiel von abdominalen Aortenaneurysmen.
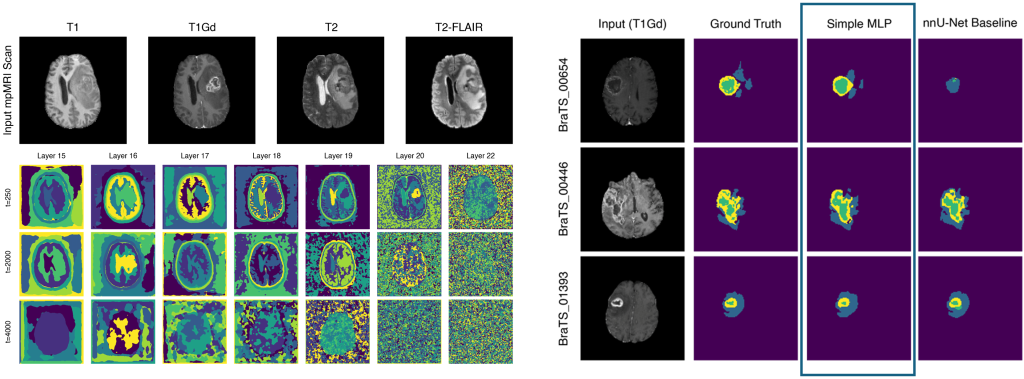
Gelernte visuelle Repräsentationen des BRATS Datensatzes für ein Diffusionsmodell (links) und daraus abgeleitete Segmentierungen eines Gehirntumors für nur zehn Trainingsbilder (rechts).
Learned Visual Representations of the BRATS dataset from Diffusion (left) and derived segmentations for only ten training slices (right).
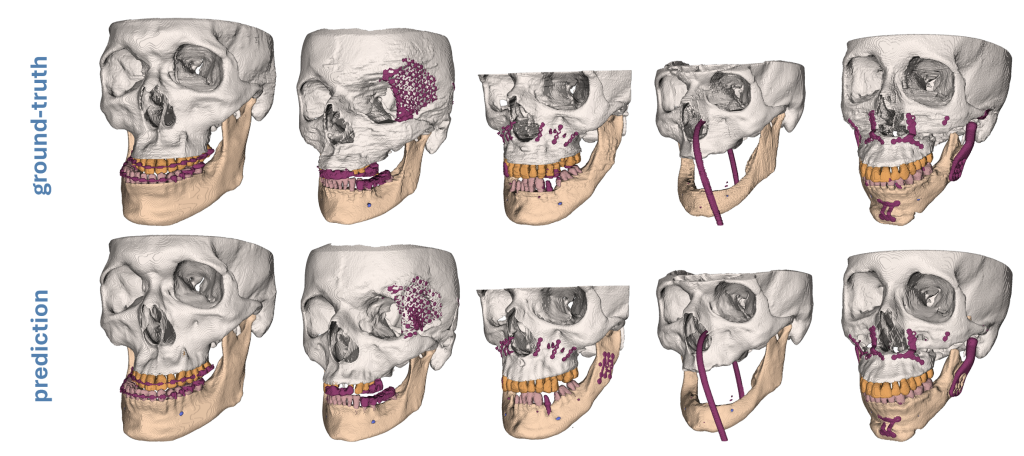
Deep Learning basierte Segmentierung von Unterkiefer, Gesichtsschädel, Zähnen, Kieferkanal und Metallstrukturen als Grundlage für patient*innenspezifisches Implantatdesign.
Medizinische Modellierung und Simulation
Unter Modellierung versteht man die vereinfachte Abbildung der Realität. Im medizinischen Kontext sind Modelle stark an klinischer Relevanz, Anwendbarkeit und Datenlage orientiert. Sie reichen von 3D-Körperoberflächenmodellen bis zu Blutflussmodellen.
In Bereichen wie Verbrennungsmedizin, chronischem Wundmanagement oder Forensik spielt die Körperoberfläche eine zentrale Rolle. Deshalb erstellt das Forschungsteam virtuelle 3D-Avatare von Patient*innen, die reale Körper möglichst exakt abbilden. Damit lassen sich Wunden präzise lokalisieren, dokumentieren und im Zeitverlauf objektiv bewerten.
Mit der biomechanischen Simulation bildet das Team Abläufe im menschlichen Organismus nach – beispielsweise den Blutfluss. Dafür nutzt es segmentierte Bilddaten, erstellt Rechengitter (Meshes) und berechnet Materialeigenschaften wie Elastizität oder Viskosität. Auf dieser Basis können Ärzt*innen sowohl geometrische als auch simulationsbasierte Merkmale bestimmen. Dazu zählen etwa Gefäßdurchmesser oder mechanische Spannungen in Gefäßwänden.
Diese Ergebnisse erleichtern Diagnosen und Behandlungsentscheidungen. Zum Beispiel lässt sich das Rupturrisiko eines Aneurysmas berechnen oder die Wirksamkeit von Eingriffen im Blutgefäßsystem beurteilen. Außerdem finden die Modelle in medizinischen Simulatoren Anwendung, um Eingriffe realitätsnah zu trainieren.
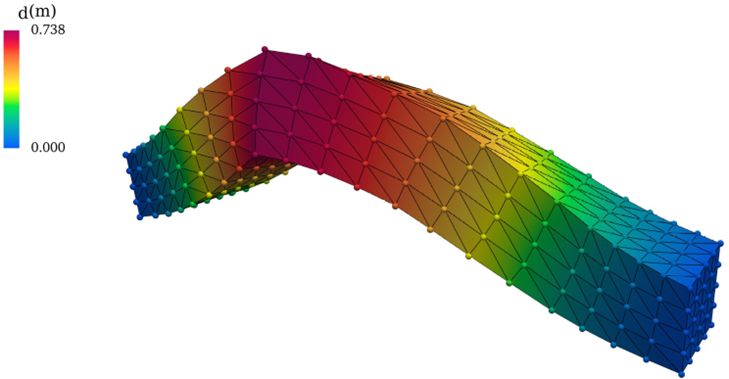
Deformierter Balken als Beispiel für ein ML-basiertes Surrogat-Modell (Perceiver-IO) zur Lösung von Elastizitätsgleichungen. Die Punkte entsprechen den Surrogat-Vorhersagen, während die durchgezogenen Linien aus der exakten numerischen Simulation (FEM) stammen.
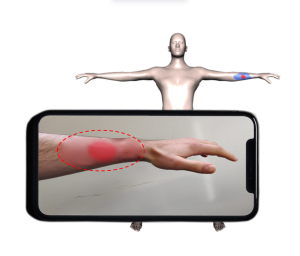
Übertragung einer Brandwunde von einem 2D Bild auf ein 3D Patient*innen-Modell. Die automatische Wundlokalisation und Anpassung des 3D Modells an die Körperform desder Patient*in ermöglicht eine objektivere Bestimmung der Wundgröße und Dokumentation des Heilungsverlaufs.
Medizinische Datenanalyse und Vorhersage
Ein wesentlicher Erfolgsfaktor für die Anwendung von Machine Learning in der Medizin ist das Vertrauen von Ärzt*innen und Patient*innen in die Datenbasis und die davon abgeleiteten Vorhersagemodelle. Grundlage für dieses Vertrauen sind Methoden zur Validierung der Daten, zur Interpretation der Modellvorhersagen und zur Analyse von Abweichungen. Durch die Bereitstellung und Nutzbarmachung dieser Methoden in einem Datenaufbereitungs- und Analyseframework, welches die vollständige Datenverarbeitungspipeline umfasst, unterstützt die Forschungsabteilung Medizin-Informatik die Mediziner*innen in der klinischen Praxis.
Die Erarbeitung der erforderlichen Strukturen zur Datenverarbeitung erfolgt im Rahmen von ausgewählten Beispielszenarien. Anwendungsbereiche sind etwa das Verlegungsmanagement auf der Intensivstation, die Optimierung des Manchester-Triage-Systems in der Notaufnahme, die optimale Applikation von Blutkonserven oder die Vorhersage kardialer Instabilität.
Im Fokus steht die Erforschung einer generischen und einfach konfigurierbaren Datenverarbeitungskette für bewährte und neueste Methoden, um auch zukünftigen Anforderungen gerecht zu werden. Neben strukturierten Daten dienen auch Bild-, Video- und Signaldaten sowie Kombinationen unterschiedlicher Datenmodalitäten und eine spezialisierte Merkmalsextraktion als Informations- und Datengrundlage.
Der Forschungsfokus liegt auf der interaktiven Datenanalyse unterschiedlicher Modalitäten mit Fokus auf Interpretierbarkeit und Rückverfolgbarkeit von Einzeldaten oder Datengruppen im Sinne von „Explainable AI“. Gerade in der Medizin ist die Transparenz für die Akzeptanz KI-basierter Software von besonders hoher Bedeutung.
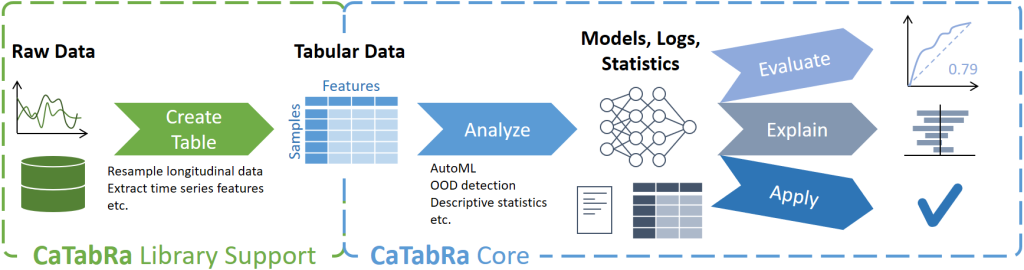
CaTabRa ist ein Python-Package zur weitgehend automatisierten Analyse von tabellarischen Daten. Dies umfasst die Erstellung von deskriptiven Statistiken, die Erstellung von Out-of-Distribution-Detektoren, das Training von Vorhersagemodellen für Klassifizierungs- und Regressionsaufgaben und die Auswertung/Erklärung/Anwendung dieser Modelle auf ungesehene Daten.
Zusammenspiel dieser Forschungsfelder
Die Forschungsfelder der Abteilung Medizin-Informatik stehen in einem sehr engen Zusammenhang. Medizinische Bilddaten bilden oftmals die Grundlage für die Modellierung, Modelle bilden wiederum die Grundlage für die medizinische Bildverarbeitung und Informationsextraktion sowie für die Simulation von Abläufen im menschlichen Körper. Auch die verwendeten Basistechnologien und -methoden der Forschungsfelder zeigen vielfältige Überlappungen. GPU-basierte (Graphics Processing Unit) parallele Berechnungen haben in den letzten Jahren den Siegeszug von Deep Learning in der Bildverarbeitung ermöglicht und stellen gleichzeitig die Basis für die Simulation von Abläufen im menschlichen Körper dar.
Physiologische Interaktionen bedürfen jedoch entsprechender Modelle von anatomischen Strukturen, welche mittels Segmentierungsmethoden aus medizinischen Bilddaten extrahiert werden. Die Registrierung – also die Errechnung einer Transformation, welche mehrere Datensätze (Modell, Bild, Volumen) in geometrische Übereinstimmung bringt – ermöglicht die Nutzung mehrerer Datenquellen sowie den Transfer von Informationen zwischen verschiedenen Datendomänen. Die Informationsextraktion erfolgt in allen Forschungsfeldern in unterschiedlicher Weise. Die folgenden medizinischen Anwendungsbeispiele verdeutlichen den Zusammenhang dieser Forschungsfelder:
Rupturrisiko von Aneurysmen
Aneurysmen werden in CTA-Scans (Computertomographie-Angiographie) diagnostiziert. Die Segmentierung von Aneurysmen und Blutgefäßen wird für die Erstellung eines Volumenmodells (Mesh) zur Blutflusssimulation verwendet. Die Simulation ermöglicht die Berechnung von Druck und Gefäßspannung. Für eine Patient*innen-Kohorte (z.B. Aneurysmenpatient*innen der letzten zehn Jahre) können diese Merkmale genutzt werden, um anhand von Machine Learning basierter Datenanalyse das Rupturrisiko zu ermitteln und darauf aufbauend eine geeignete Behandlungsstrategie zu wählen.
Verbrennungsklassifikation
Patient*innen mit Verbrennungswunden werden in der Notaufnahme initial behandelt. Mittels medizinischer Modellierungsmethoden wird ein virtuelles 3D-Körperoberflächenmodell anhand eines RGB-D-Scans an die Patient*innen angepasst. Die Verbrennungstiefe wird anhand von Bildanalysemethoden klassifiziert. Die Wunden (Ausdehnung und Tiefe) werden am Körperoberflächenmodell dokumentiert. Der zeitliche Verlauf der Wunde aufgrund der späteren Behandlung wird am Körperoberflächenmodell dokumentiert und kann so für die Verbesserung der Behandlung zukünftiger Patient*innen genutzt werden.
Das gemeinsame Ziel aller Arbeiten ist die weitere Verbreitung von individualisierter und evidenzbasierter Medizin. Dazu müssen aktuelle Methoden aus der Forschung frühzeitig mit medizinischen Fachexpert*innen weiterentwickelt werden. Nur so kann sichergestellt werden, dass mittelfristig aktuelle Methoden zum Wohle der Patient*innen auch in der klinischen Praxis eingesetzt werden.
Dieses Projekt wird aus Forschungsförderungsmitteln des Landes Oberösterreich finanziert. Die RISC Software GmbH ist Teil des Netzwerkes der UAR (Upper Austrian Research).
Projektpartner


Details zum Projekt
- Projekt-Kurztitel: MIMAS.ai
- Projekt-Langtitel: Medical Image Processing, Modeling and Simulation based on Artificial Intelligence
- Fördercall: Programm zur Stimulierung der Erschließung/Erweiterung von zukunftsweisenden Forschungsfeldern bei den Oö. außeruniversitären Forschungseinrichtungen im Zeitraum 01.01.2022 – 31.12.2029
- Projektpartner*innen:
- RISC Software GmbH, Forschungsabteilung Medizin-Informatik
- Budgetvolumen (gesamt): 2,398 Mio. Euro
- davon Förderung (gesamt): 2,398 Mio. Euro
- Laufzeit: 01/2022 – 12/2025
Ansprechpartner

Dr. Michael Giretzlehner
Head of Research Unit Medical Informatics